M/L Classification : Wine 품종 분류하기
1차로 13개 feature 전부를 대상으로 RandomForest Classification 과 DecisionTree 알고리즘으로 모델을 생성하고, 모델의 적합성을 검증해 봅니다.
Contents
데이터 준비
데이터 다운로드 및 분리
출처 : UCI Machine Learning Repository 의 WINE 데이터
13개의 feature / 178 records wine 데이터
1) Alcohol
2) Malic acid
3) Ash
4) Alcalinity of ash
5) Magnesium
6) total phenols
7) Flavanoids
8) Nonflavanoid phenols
9) Proanthocyanins
10) Color intensity
11) Hue
12) OD280/OD315 of diluted wines
13) Proline
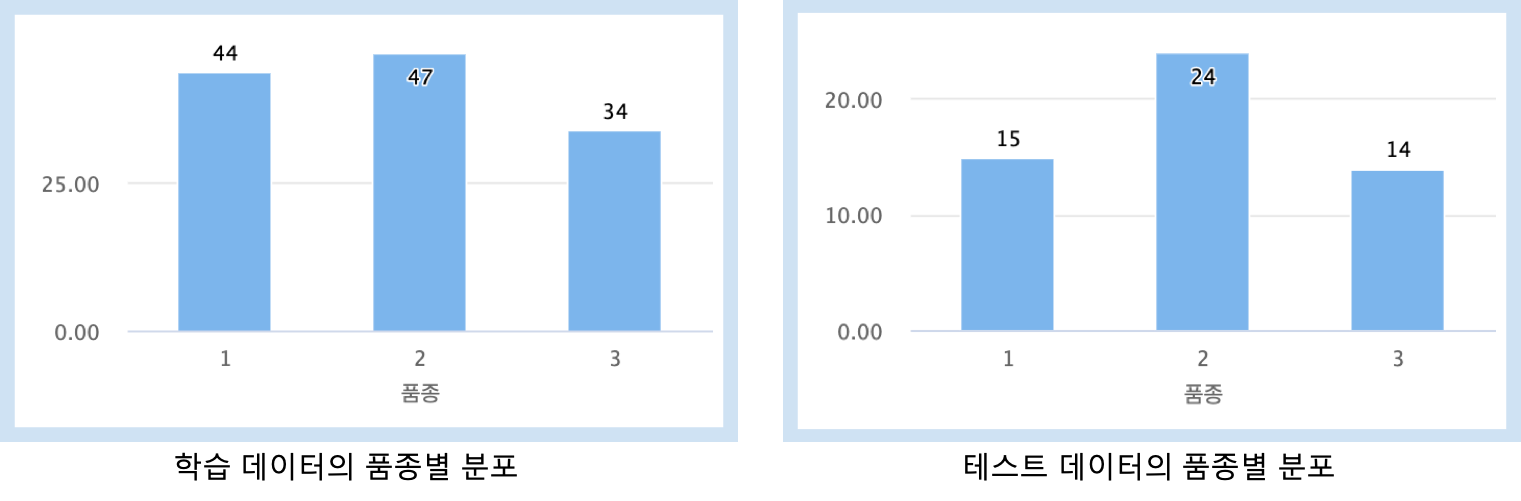
- 로컬PC 에 다운받은 wine 데이터를 70% 학습데이터, 30% 테스트 데이터로 분리합니다.
178개의 레코드를 랜덤 추출 방식으로 학습데이터와 테스트데이터를 분리하여 저장합니다.
Train_wine_data.csv (125 rows) , Test_wine_data.csv (53 rows)
- 학습데이터와 테스트데이터 파일을 IRIS 의 HDFS브라우저 메뉴의 MinIO 를 선택하여 업로드합니다.
MinIO 에 데이터 업로드하기 : MINIO 에 업로드하기
데이터모델 만들기 - MinIO
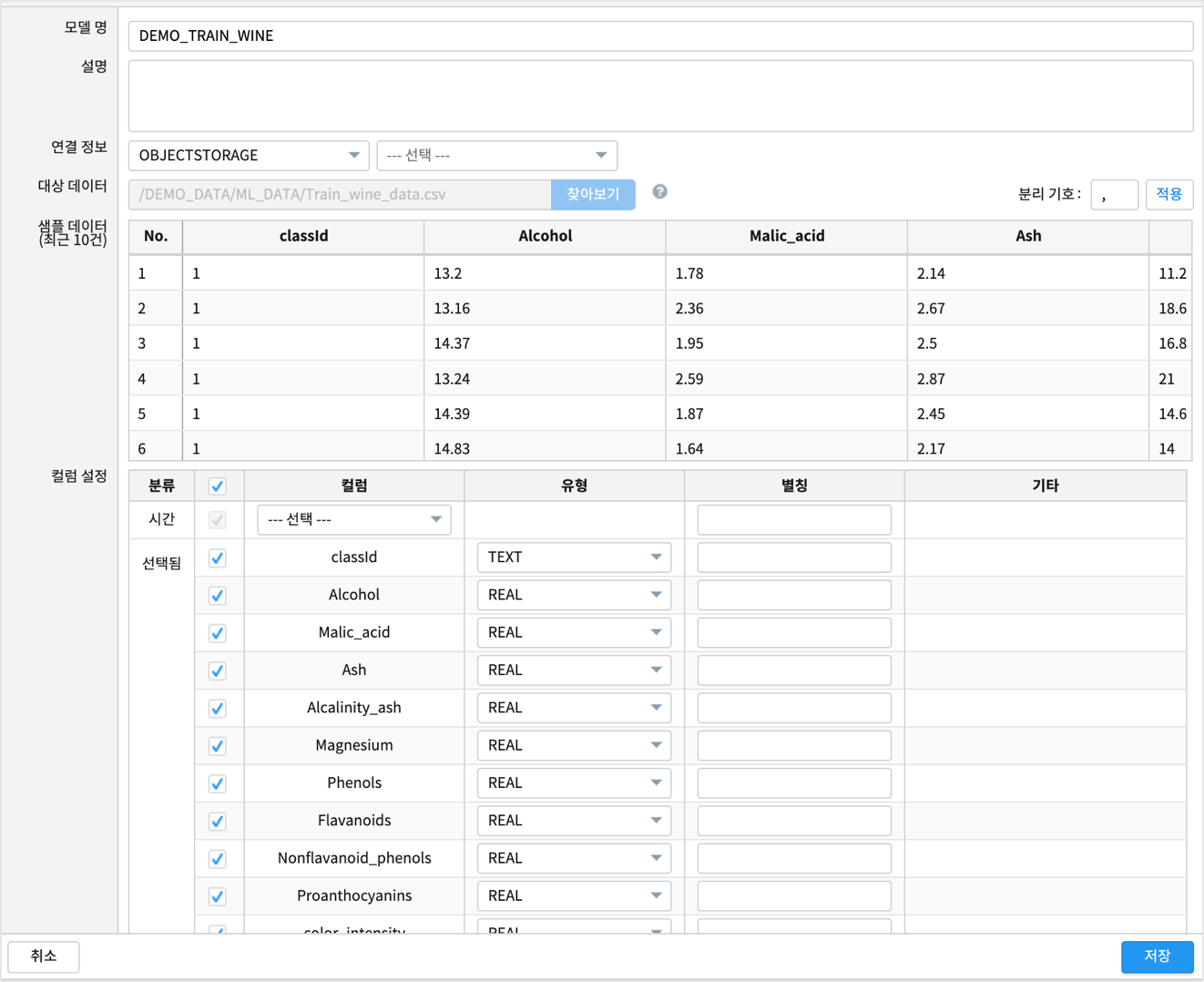
- 데이터모델 메뉴에서 MinIO 에 저장된 Train_wine_data.csv, Test_wine_data.csv 를 데이터모델로 생성합니다.
DEMO_TRAIN_WINE , DEMO_TEST_WINE
숫자형 데이터는 데이터 타입을 TEXT 에서 REAL 로 변경하여 데이터 모델을 생성합니다.
Machine Learining 모델 만들기
데이터 전처리
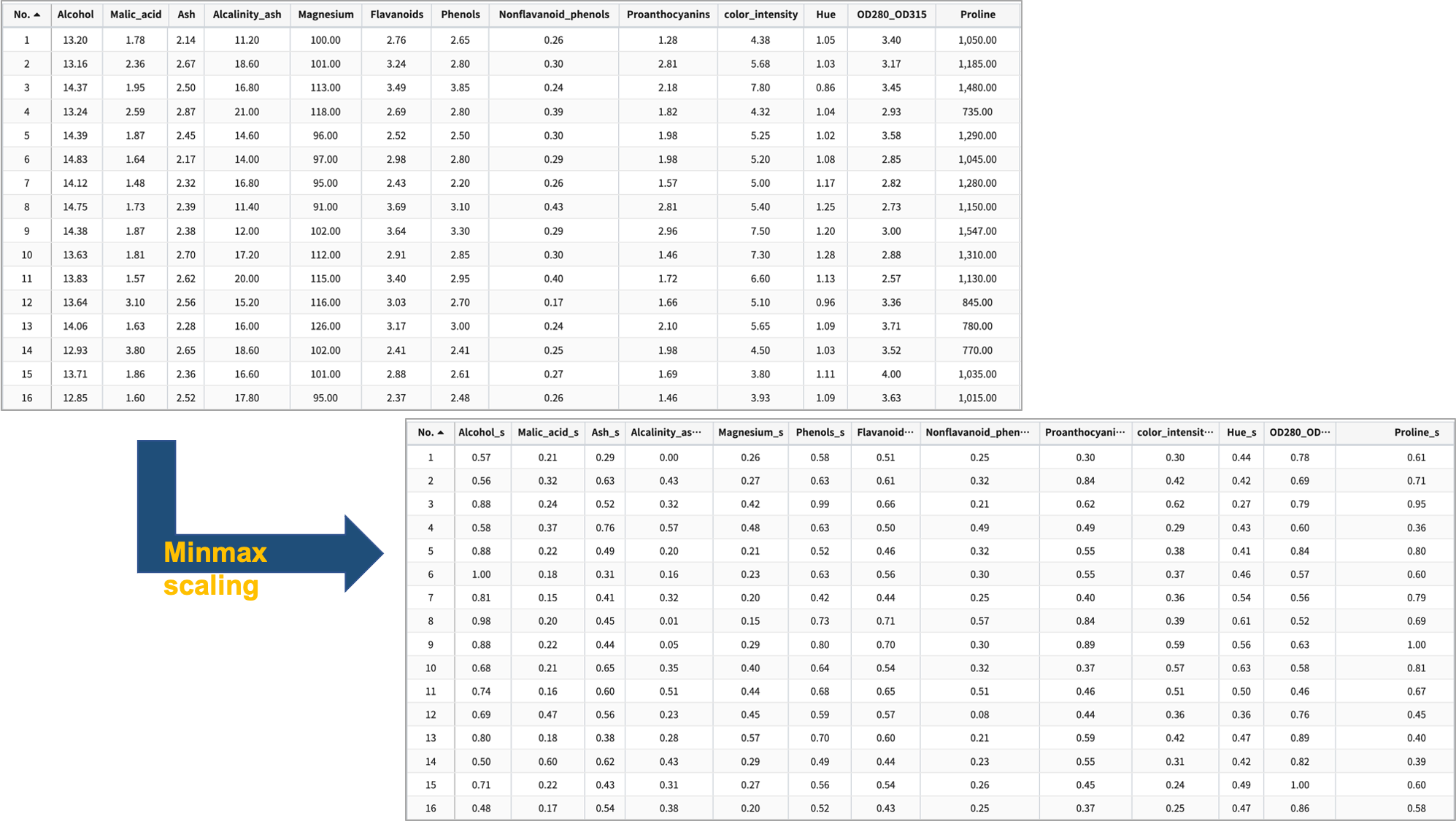
wine 별로 측정한 13개의 feature 데이터를 스케일링 합니다. 여기서는 minmax scaling 을 사용합니다.
사용 command : scaler
데이터 스케일링은 데이터 전처리 과정의 하나로 모델 알고리즘 학습에서 중요한 부분입니다.
데이터 스케일링을 해주는 이유는 데이터의 값이 너무 크거나 작은 경우에는 학습과정에서 0으로 수렴하거나 무한으로 발산해버릴 수 있기 때문입니다.
현재 구현된 스케일링은 standard, minmax 가 있습니다.
1) Standard 스케일러
각 feature의 평균을 0, 분산을 1로 변경합니다. 모든 특성들이 같은 스케일을 갖게 됩니다.
2) MinMax 스케일러
모든 feature가 0과 1사이에 위치하도록 만듭니다. 데이터가 2차원 셋일 경우, 모든 데이터는 x축의 0과 1 사이에, y축의 0과 1사이에 위치하게 됩니다.
검색 command 예시
* | scaler minmax Alcohol to Alcohol_s, Malic_acid to Malic_acid_s. Ash to Ash_s
원본 데이터와 minmax 스케일링 한 데이터 예시
RandomForest classification 모델 학습
- 사용 Commandfit
fit 에 사용한 RandomForest Classification
13개 feature 의 값으로 포도 품종에 해당하는 컬럼인 classId 를 분류하는 분류 모델을 RandomForest classification 알고리즘으로 만들어 봅니다.
- RandomForest Classification( 랜덤포레스트 위키설명 ) 은 앙상블(앙상블 학습법 위키 설명) 머신러닝 모델의 하나입니다.
다수의 의사결정 트리를 만들고, 그 나무들의 분류를 취합하여 최종적으로 결론을 도출하는 방식입니다.
다수의 나무를 기반으로 예측하므로, 오버피팅 등의 영향력이 줄어드는 효과를 볼 수 있습니다.
검색 명령어 창에서 실행하는 Command 예시
* | scaler minmax Alcohol to Alcohol_s,
Malic_acid to Malic_acid_s, Ash to Ash_s,
Alcalinity_ash to Alcalinity_ash_s,
Magnesium to Magnesium_s, Phenols to Phenols_s,
Flavanoids to Flavanoids_s, Nonflavanoid_phenols to Nonflavanoid_phenols_s,
Proanthocyanins to Proanthocyanins_s, color_intensity to color_intensity_s,
Hue to Hue_s, OD280_OD315 to OD280_OD315_s, Proline to Proline_s
| fit RandomForestClassification
FEATURES
Alcohol_s,Malic_acid_s,Ash_s, Alcalinity_ash_s,Magnesium_s,
Phenols_s,Flavanoids_s, Nonflavanoid_phenols_s,Proanthocyanins_s,color_intensity_s,Hue_s, OD280_OD315_s,Proline_s
LABEL classId maxDepth=20
INTO DEMO_02_RF_CLASSIFICATION_WINE
command 의 의미
13개 feature 를 minmax 스케일링으로 전처리하고 RandomForestClassification 알고리즘으로 fit
- FEATURE 는 13개의 스케일링 변환된 컬럼
- LABEL 은 품종을 나타내는 classId 컬럼
- fit 으로 학습된 모델은 DEMO_02_RF_CLASSIFICATION_WINE 이라는 모델이름으로 저장
- IRIS Analyzer 의 검색 메뉴에서 분석 탬플릿 인 DEMO_RF_분류_와인_TRAIN 이 배포되어 있습니다.
학습용 wine데이터 모델과 모델 생성 code 가 같이 저장되어 있습니다. 더블클릭하여 검색 메뉴로 불러오기를 할 수 있습니다.
모델 결과는 동일한 이름을 사용할 수 없으므로 그대로 실행하면 에러가 발생합니다.
fit 으로 새 모델을 생성하려면 DEMO_02_RF_CLASSIFICATION_WINE 가 아닌 다른 모델 이름으로 수정해서 실행하시기 바랍니다.
모델 평가
생성한 모델의 성능을 평가하는 지표들이 화면에 같이 출력됩니다.
모델 평가 용어 정의
TP (True Positive) : 참을 참으로 정확하게 예측
TN (True Negative) : 참을 거짓으로 예측
FP (False Positive) : 거짓을 참으로 예측
FN (False Negative) : 거짓을 거짓으로 정확하게 예측
정확도(accuracy)는 전체 샘플 중 맞게 예측한 샘플 수의 비율을 뜻한다.
높을수록 좋은 모형이다.
accuracy = (TP + TN) / (TP + TN + FP + FN)
정밀도(precision)은 양성 클래스에 속한다고 출력한 샘플 중 실제로 양성 클래스에 속하는 샘플 수의 비율을 말한다.
높을수록 좋은 모형이다. 1번 품종으로 예측한 와인이 실제로 1번 품종인 레코드의 비율이다.
precision = TP / (TP + FP)
재현율(recall)은 실제 양성 클래스에 속한 표본 중에 양성 클래스에 속한다고 출력한 표본의 수의 비율을 뜻한다.
높을수록 좋은 모형이다.
TPR(true positive rate) 또는 민감도(sensitivity)라고도 한다.
recall = TP / ( TP + FN)
F-Score 는 재현율의 가중조화평균(weight harmonic average)을 말한다. 정밀도에 주어지는 가중치를 베타(beta)라고 한다.
베타가 1인 경우를 특별히 F1 점수 라고 한다.
F1 = 2 * precision * recall / (precision + recall)
참고) 조화평균은 측정값의 역수를 합한 값으로 평균을 구한 값. 샘플의 수가 집단별로 동일하지 않을 때 적용하며,
극단적인 값의 영향력을 줄이기 위해 사용되곤 합니다.
fit 명령어 실행 결과로 정확도(accuracy), 정밀도(precision), 재현율(recall), F1 값을 모델의 성능 지표로 출력합니다.
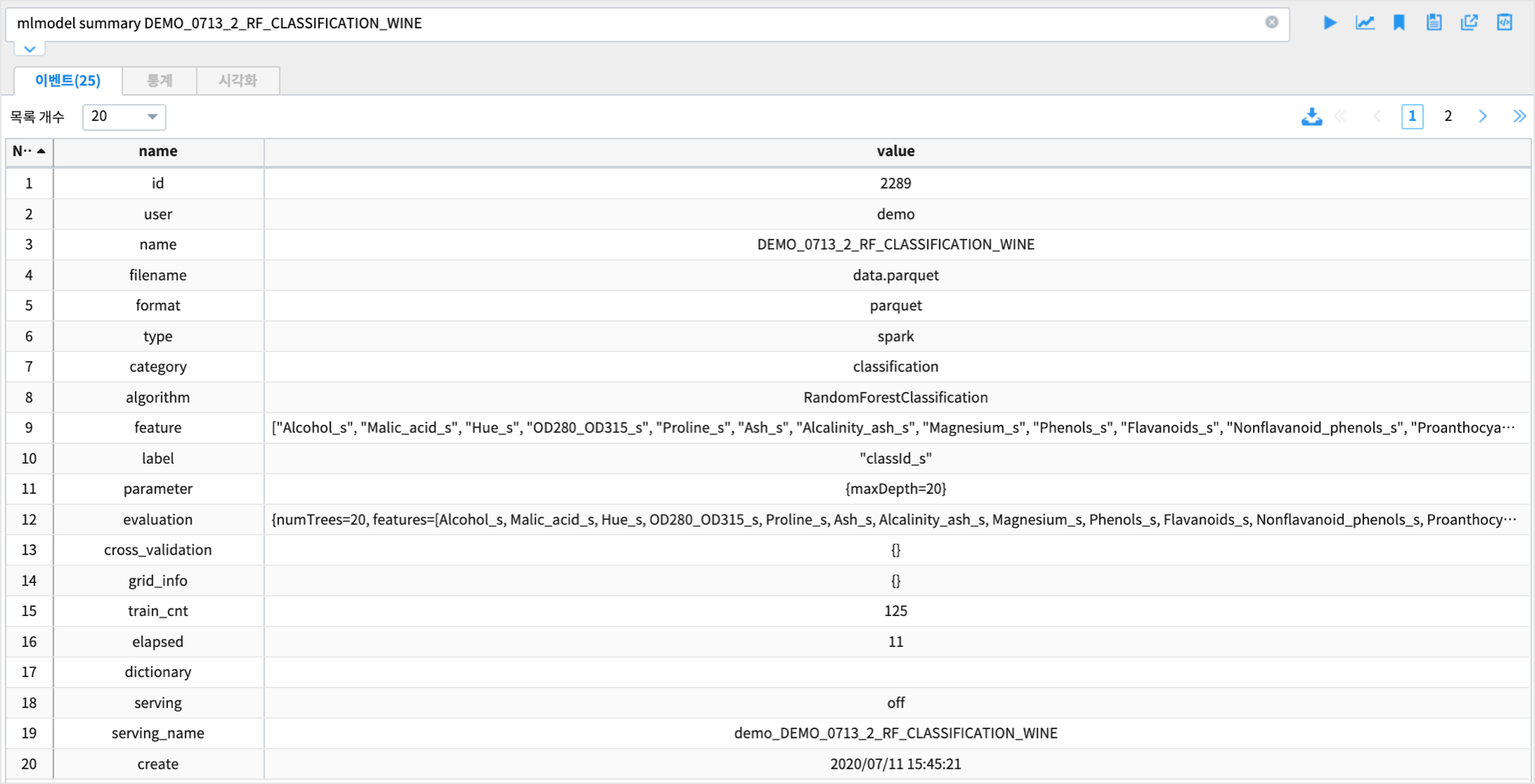
생성한 Machine Learning 모델은 mlmodel 명령어로 조회할 수 있습니다.
mlmodel summary DEMO_02_RF_CLASSIFICATION_WINE
테스트 데이터의 품종 예측하기
검색 명령어 창에서 실행하는 Command 예시
* | scaler minmax Alcohol to Alcohol_s,
Malic_acid to Malic_acid_s,
Ash to Ash_s,
Alcalinity_ash to Alcalinity_ash_s,
Magnesium to Magnesium_s,
Phenols to Phenols_s,
Flavanoids to Flavanoids_s,
Nonflavanoid_phenols to Nonflavanoid_phenols_s,
Proanthocyanins to Proanthocyanins_s,
color_intensity to color_intensity_s,
Hue to Hue_s,
OD280_OD315 to OD280_OD315_s,
Proline to Proline_s
| predict DEMO_02_RF_CLASSIFICATION_WINE
Alcohol_s,Malic_acid_s, Ash_s,
Alcalinity_ash_s, Magnesium_s, Phenols_s,
Flavanoids_s, Nonflavanoid_phenols_s, Proanthocyanins_s,
color_intensity_s, Hue_s, OD280_OD315_s, Proline_s
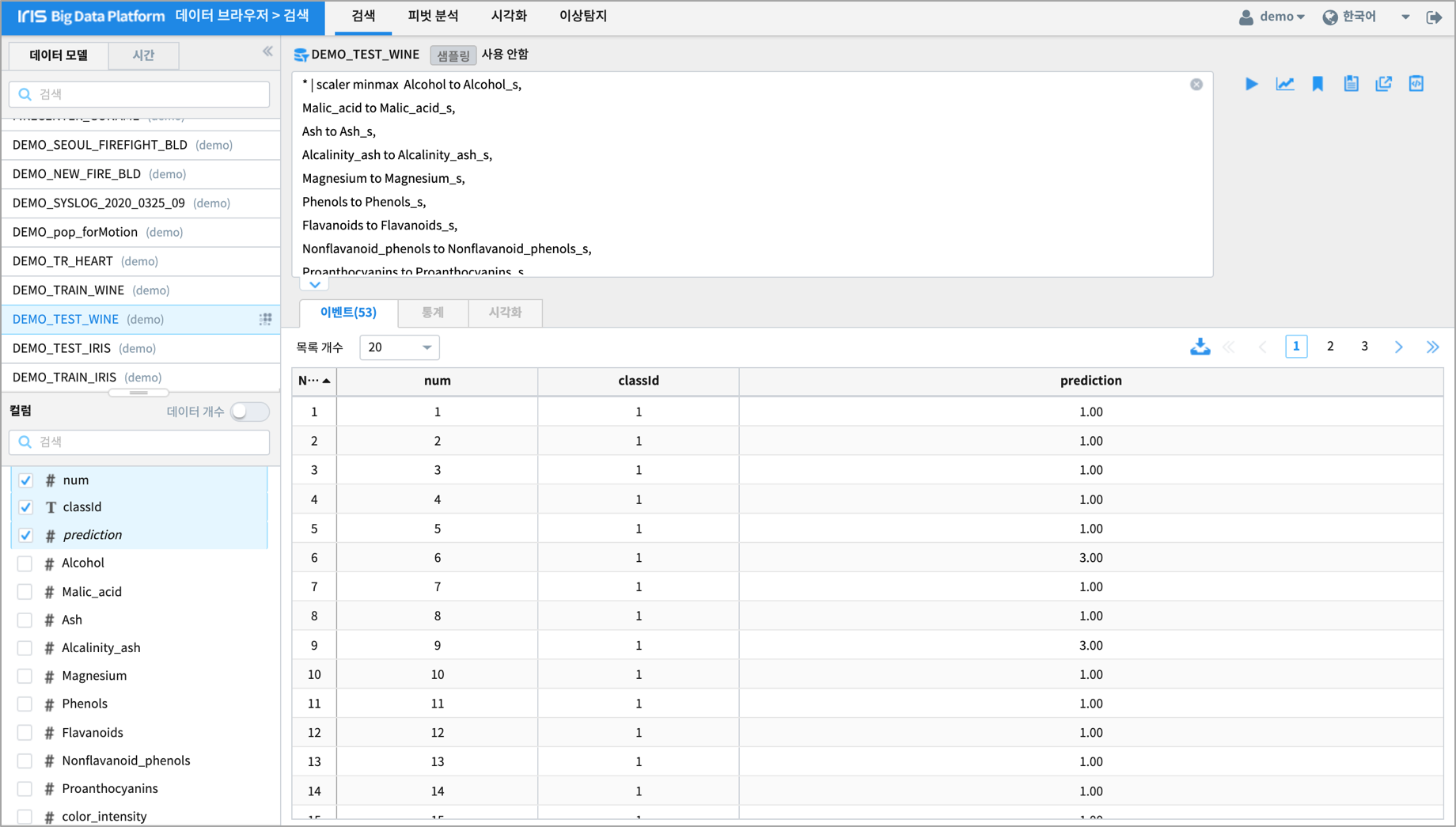
예측 결과 분석
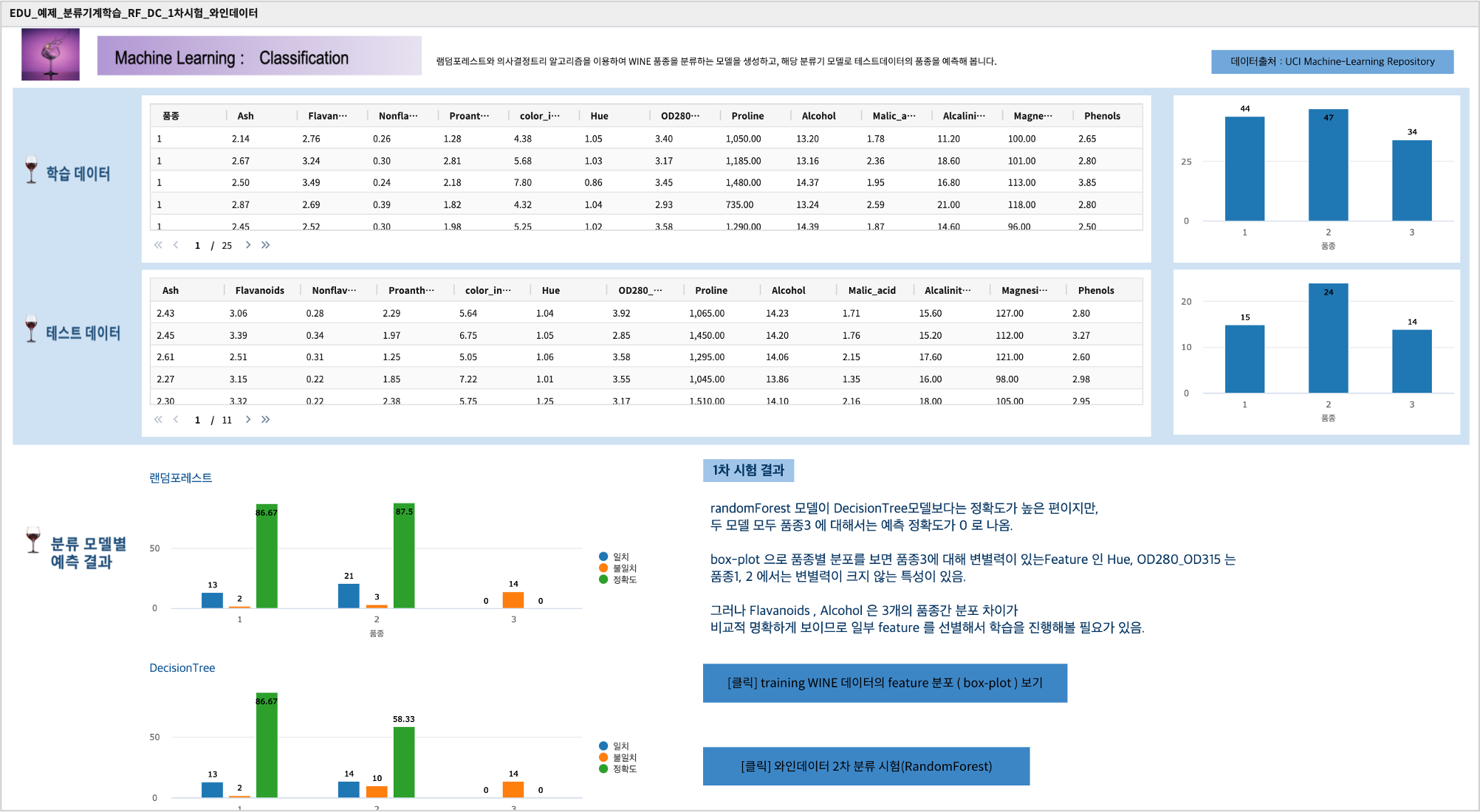
테스트 데이터에서 품종 3번은 14개 와인 모두 예측을 하지 못했습니다.
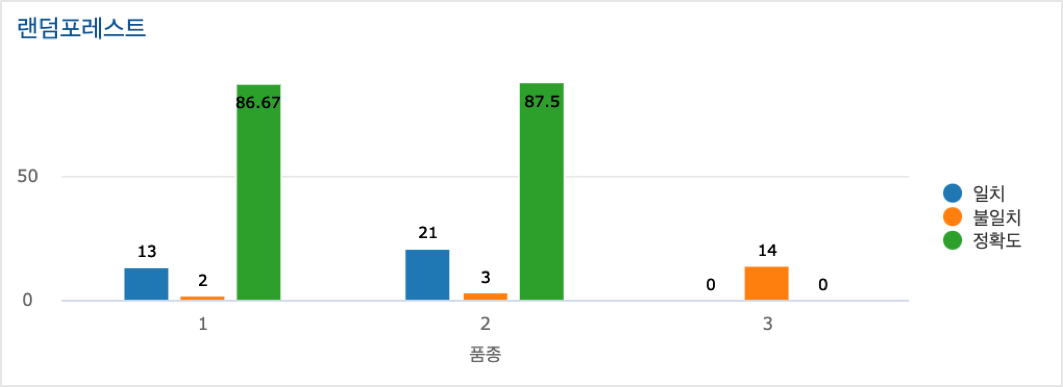
원인을 알아보고 더 성능 좋은 모델을 만들기 위해서는, 정확도 높은 모델이 나올 때 까지 2차, 3차 학습 등 1차 학습과 비슷한 과정들이 추가로 필요합니다.
참고 : 보고서 분류_RF_DC_1차시험_와인데이터
분류_RF_DC_1차시험_와인데이터 는 테스트 데이터를 RandomForest 와 DecisionTree 모델로 각각 예측한 결과를 bar-chart 로 그리고, 13개 feature 의 분포를 그린 box-plot 들을 링크로 만든 보고서 입니다.